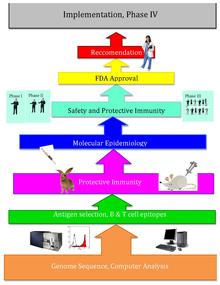
Omvendt vaccinologi - Reverse vaccinology
Omvendt vaccinologi er en forbedring af vaccinologi, der anvender bioinformatik, som var banebrydende af Rino Rappuoli og først blev brugt mod Serogroup B meningococcus . Siden da er det blevet brugt på flere andre bakterievacciner.
Computational tilgang
Grundtanken bag omvendt vaccinologi er, at et helt patogent genom kan screenes ved hjælp af bioinformatikmetoder til at finde gener. Nogle træk, som generne overvåges for, kan indikere antigenicitet og omfatte gener, der koder for proteiner med ekstracellulær lokalisering , signalpeptider og B -celleepitoper . Derefter filtreres disse gener efter ønskelige attributter, der ville gøre gode vaccinemål, såsom ydre membranproteiner . Når kandidaterne er identificeret, produceres de syntetisk og screenes i dyremodeller af infektionen.
Historie
Efter at Craig Venter offentliggjorde genomet for den første fritlevende organisme i 1995, blev genomer fra andre mikroorganismer lettere tilgængelige i slutningen af det tyvende århundrede. Omvendt vaccinologi, design af vacciner ved hjælp af patogenets sekventerede genom, stammer fra denne nye rigdom af genomisk information samt teknologiske fremskridt. Omvendt vaccinologi er meget mere effektiv end traditionel vaccinologi, som kræver voksende store mængder af specifikke mikroorganismer samt omfattende vådlaboratorietest.
I 2000 udviklede Rino Rappuoli og J. Craig Venter Institute den første vaccine ved hjælp af Reverse Vaccinology mod Serogroup B meningococcus. J. Craig Venter Institute og andre fortsatte derefter arbejdet med vacciner mod A Streptococcus, B Streptococcus, Staphylococcus aureus og Streptococcus pneumoniae.
Omvendt vaccinologi med Meningococcus B
Forsøg på omvendt vaccinologi begyndte først med Meningococcus B (MenB). Meningococcus B forårsagede over 50% af meningokok meningitis, og forskere havde ikke været i stand til at oprette en vellykket vaccine til patogenet på grund af bakteriens unikke struktur. Denne bakteries polysaccharidskal er identisk med den for et menneskeligt selvantigen, men dens overfladeproteiner varierer meget; og manglen på information om overfladeproteinerne medførte, at udviklingen af en vaccine var ekstremt vanskelig. Som et resultat vendte Rino Rappuoli og andre forskere mod bioinformatik for at designe en funktionel vaccine.
Rappuoli og andre ved J. Craig Venter Institute sekventerede først MenB -genomet. Derefter scannede de det sekventerede genom for potentielle antigener. De fandt over 600 mulige antigener, som blev testet ved ekspression i Escherichia coli . De mest universelt anvendelige antigener blev anvendt i prototypevaccinerne. Flere viste sig at fungere med succes hos mus, men disse proteiner alene interagerede ikke effektivt med det menneskelige immunsystem på grund af ikke at fremkalde et godt immunrespons for at beskyttelsen kunne opnås. Senere ved tilsætning af ydre membranvesikler, der indeholder lipopolysaccharider fra oprensning af blebs på gramnegative kulturer. Tilsætningen af denne adjuvans (tidligere identificeret ved anvendelse af konventionelle vaccinologimetoder) øgede immunresponsen til det niveau, der var påkrævet. Senere viste vaccinen sig at være sikker og effektiv hos voksne mennesker.
Efterfølgende forskning i omvendt vaccinologi
Under udviklingen af MenB -vaccinen vedtog forskere de samme metoder til omvendt vaccinologi for andre bakterielle patogener. A Streptococcus og B Streptococcus vacciner var to af de første Reverse Vaccines, der blev oprettet. Fordi disse bakteriestammer fremkalder antistoffer, der reagerer med humane antigener, skulle vaccinerne for disse bakterier ikke indeholde homologier med proteiner, der er kodet i det humane genom for ikke at forårsage bivirkninger, og dermed fastslå behovet for genombaseret omvendt vaccinologi.
Senere blev Reverse Vaccinology brugt til at udvikle vacciner til antibiotikaresistente Staphylococcus aureus og Streptococcus pneumoniae
Fordele og ulemper
Den største fordel ved omvendt vaccinologi er at finde vaccinemål hurtigt og effektivt. Traditionelle metoder kan tage årtier at opklare patogener og antigener, sygdomme og immunitet. Imidlertid kan In silico være meget hurtig, hvilket gør det muligt at identificere nye vacciner til test inden for få år. Ulempen er, at kun proteiner kan målrettes ved hjælp af denne proces. Hvorimod konventionelle vaccinologimetoder kan finde andre biomolekylære mål, såsom polysaccharider .
Tilgængelig software
Selvom brug af bioinformatisk teknologi til at udvikle vacciner er blevet typisk i de sidste ti år, har generelle laboratorier ofte ikke den avancerede software, der kan gøre dette. Imidlertid er der et stigende antal programmer, der gør oplysninger om omvendt vaccinologi mere tilgængelige. NERVE er et relativt nyt databehandlingsprogram. Selvom den skal downloades og ikke indeholder alle forudsigelser om epitoper, hjælper den dog med at spare tid ved at kombinere beregningstrinene med reverse vaccinologi til ét program. Vaxign, et endnu mere omfattende program, blev oprettet i 2008. Vaxign er webbaseret og fuldstændig offentligt tilgængelig.
Selvom Vaxign har vist sig at være ekstremt præcist og effektivt, anvender nogle forskere stadig onlinesoftwaren RANKPEP til forudsigelser af peptidbinding. Både Vaxign og RANKPEP anvender PSSM'er (Position Specific Scoring Matrices), når de analyserer proteinsekvenser eller sekvensjusteringer.
Computerstøttet bioinformatik-projekter bliver ekstremt populære, da de hjælper med at guide laboratorieeksperimenterne.
Andre udviklinger på grund af omvendt vaccinologi og bioinformatik
- Omvendt vaccinologi har forårsaget et øget fokus på patogen biologi.
- Omvendt vaccinologi førte til opdagelsen af pili i grampositive patogener som A streptococcus, B streptococcus og pneumococcus. Tidligere troede man, at alle grampositive bakterier ikke havde nogen pili.
- Omvendt vaccinologi førte også til opdagelsen af faktor G -bindende protein i meningococcus, som binder til komplement faktor H hos mennesker. Binding til komplementfaktoren H giver meningokokker mulighed for at vokse i humant blod, mens de blokerer alternative veje. Denne model passer ikke til mange dyrearter, som ikke har den samme komplementfaktor H som mennesker, hvilket indikerer differentiering af meningokokker mellem forskellige arter.
Referencer
- ^ Pizza et al. Identifikation af vaccinkandidater mod Serogruppe B Meningococcus ved Whole-Genome Sequencing Science 2000 287: 1816-1820
- ^ Rappuoli, Rino. Reverse Vaccinology Current Opinion in Microbiology 2000, 3: 445–450
- ^ CH Woelk, et al. "Forbedring af omvendt vaccinologi med en machine learning -tilgang." Vaccine 29, nr. 45 (nd): 8156-8164. Science Citation Index, EBSCOhost (åbnet 30. september 2012).
- ^ Michalik, M., Djahanshiri, B., Leo, JC, & Linke, D. (2016). Omvendt vaccinologi: Vejen fra genomer og epitopforudsigelser til skræddersyede rekombinante vacciner. Metoder i molekylærbiologi , 1403, 87–106. https://doi.org/10.1007/978-1-4939-3387-7_4
- ^ a b c d e f g h Alessandro S, Rino R. Anmeldelse: Reverse Vaccinology: Developing Vaccines in the Era of Genomics. Immunitet [seriel online]. nd; 33: 530-541. Tilgængelig fra: ScienceDirect , Ipswich, MA. Adgang til 30. september 2012.
- ^ Rappuoli, R. & A. Aderem. 2011. En 2020 -vision for vacciner mod hiv, tuberkulose og malaria. Natur 473: 463.
- ^ He Y, Xiang Z, Mobley H. Vaxign: det første webbaserede vaccinedesignprogram til omvendt vaccinologi og applikationer til vaccineudvikling. Journal of Biomedicine & Biotechnology [seriel online]. 2010; Tilgængelig fra: CINAHL Plus med fuld tekst, Ipswich, MA. Adgang til 30. september 2012.
- ^ Reche PA, Glutting JP og Reinherz EL. Forudsigelse af MHC klasse I bindende peptider ved hjælp af profilmotiver. Human Immunology 63, 701-709 (2002).
- ^ Sandro V, Jennifer L. G, Francesco F, et al. Anmeldelse: Computerstøttet bioteknologi: fra immuno-informatik til omvendt vaccinologi. Trends In Biotechnology [seriel online]. nd; 26: 190-200. Tilgængelig fra: ScienceDirect, Ipswich, MA. Adgang til 30. september 2012.