Ensembl genom database -projekt - Ensembl genome database project
 | |
|---|---|
| Indhold | |
| Beskrivelse | Ensembl |
| Kontakt | |
| Forskningscenter | |
| Primær henvisning | Yates, et al. (2020) |
| Adgang | |
| Internet side | www |
Ensembl genomdatabaseprojekt er et videnskabeligt projekt ved European Bioinformatics Institute , som blev lanceret i 1999 som reaktion på den forestående gennemførelse af Human Genome Project . Ensembl sigter mod at levere en centraliseret ressource til genetikere, molekylærbiologer og andre forskere, der studerer genomerne for vores egen art og andre hvirveldyr og modelorganismer . Ensembl er en af flere velkendte genombrowsere til hentning af genomisk information.
Lignende databaser og browsere findes på NCBI og University of California, Santa Cruz (UCSC) .
Baggrund
Det menneskelige genom består af tre milliarder basepar , som koder for cirka 20.000–25.000 gener . Imidlertid er genomet alene til ringe nytte, medmindre lokaliteter og relationer mellem individuelle gener kan identificeres. En mulighed er manuel annotering , hvor et team af forskere forsøger at lokalisere gener ved hjælp af eksperimentelle data fra videnskabelige tidsskrifter og offentlige databaser. Dette er dog en langsom og omhyggelig opgave. Alternativet, kendt som automatiseret annotering, er at bruge computernes magt til at udføre den komplekse mønstertilpasning af protein til DNA .
I Ensembl -projektet føres sekvensdata ind i genannotationssystemet (en samling software "pipelines" skrevet i Perl ), som skaber et sæt forudsagte genlokationer og gemmer dem i en MySQL -database til efterfølgende analyse og visning. Ensembl gør disse data frit tilgængelige for verdens forskningssamfund. Alle data og kode produceret af Ensembl -projektet kan downloades, og der er også en offentligt tilgængelig databaseserver, der tillader fjernadgang. Derudover giver Ensembl-webstedet computergenererede visuelle visninger af meget af dataene.
Over tid har projektet udvidet til at omfatte yderligere arter (herunder de vigtigste modelorganismer såsom mus , bananflue og zebrafisk ) samt en bredere vifte af genomiske data, herunder genetiske variationer og regulatoriske funktioner. Siden april 2009 har et søsterprojekt, Ensembl Genomes , udvidet omfanget af Ensembl til hvirvelløse metazoer , planter , svampe , bakterier og protister , mens det oprindelige projekt fortsat fokuserer på hvirveldyr.
Visning af genomiske data
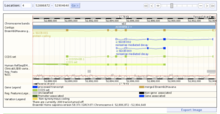
Centralt i Ensembl -konceptet er evnen til automatisk at generere grafiske visninger af tilpasningen af gener og andre genomiske data mod et referencegenom . Disse vises som dataspor, og individuelle spor kan tændes og slukkes, så brugeren kan tilpasse displayet, så det passer til deres forskningsinteresser. Interfacet gør det også muligt for brugeren at zoome ind på et område eller bevæge sig langs genomet i begge retninger.
Andre displays viser data i forskellige opløsningsniveauer, fra hele karyotyper ned til tekstbaserede repræsentationer af DNA- og aminosyresekvenser , eller præsenterer andre typer display såsom træer af lignende gener ( homologer ) på tværs af en række arter. Grafikken suppleres af tabelskærme, og i mange tilfælde kan data eksporteres direkte fra siden i en række forskellige standardfilformater, såsom FASTA .
Eksternt producerede data kan også tilføjes til displayet ved at uploade en passende fil i et af de understøttede formater, f.eks. BAM , BED eller PSL .
Grafik genereres ved hjælp af en pakke tilpassede Perl -moduler baseret på GD , standard Perl -grafikdisplaybibliotek.
Alternative adgangsmetoder
Ud over sit websted tilbyder Ensembl en REST API og en Perl API (Application Programming Interface), der modellerer biologiske objekter såsom gener og proteiner, så enkle scripts kan skrives for at hente data af interesse. Den samme API bruges internt af webgrænsefladen til at vise dataene. Det er opdelt i sektioner som core API, compara API (til komparative genomiske data), variation API (for adgang til SNP'er, SNV'er, CNV'er ..) og den funktionelle genomics API (for at få adgang til regulatoriske data). Ensembl -webstedet indeholder omfattende oplysninger om, hvordan du installerer og bruger API'et .
Denne software kan bruges til at få adgang til den offentlige MySQL -database og undgå behovet for at downloade enorme datasæt. Brugerne kunne endda vælge at hente data fra MySQL med direkte SQL -forespørgsler, men dette kræver et omfattende kendskab til det aktuelle databaseskema.
Store datasæt kan hentes ved hjælp af BioMart data-mining værktøj. Det giver en webgrænseflade til download af datasæt ved hjælp af komplekse forespørgsler.
Sidst er der en FTP -server, som kan bruges til at downloade hele MySQL -databaser samt nogle udvalgte datasæt i andre formater.
Nuværende arter
De kommenterede genomer omfatter de mest fuldstændigt sekventerede hvirveldyr og udvalgte modelorganismer. Alle er eukaryoter, der er ingen prokaryoter. Fra 2008 inkluderer dette:
-
Chordata
-
Mammalia
-
Euarchontoglires
- Primater : bushbaby , chimpanse , menneske, makak , muselemur , orangutang , tarsier ;
- SCANDENTIA : træ spidsmus ;
- Glires (= Gnavere + Lagomorphs): guineapig , kænguru rotte , mus , rotte , egern , pika , kanin ;
- Laurasiatheria : ko , delfin , alpaca , gris , kat , hund , hest , megabat , mikrobat , pindsvin , spidsmus ;
- Afrotheria: elefant , hyrax , tenrec
- Xenarthra : bæltedyr , dovendyr ;
- Marsupialia : opossum , wallaby ;
- Monotremes : næbdyr ;
-
Euarchontoglires
- Fugle : kylling , zebrafink ;
- Lepidosauria : anole firben (præ);
- Lissamphibia : Xenopus tropicalis ;
- Teleost fisk: Takifugu rubripes ( fugu ), Tetraodon nigroviridis (grøn plettet pufferfish), Danio rerio ( zebrafisk ), Oryzias latipes ( medaka ), Gasterosteus aculeatus ( stickleback );
- Cyclostomata : Petromyzon marinus ( sea lamprey ) (pre);
- Tunikaer : Ciona intestinalis , Ciona savignyi ;
-
Mammalia
- Ikke-hvirveldyr
- Insekter : Drosophila melanogaster (fruitfly), Anopheles gambiae (myg), Aedes aegypti (myg)
- Orm : Caenorhabditis elegans
- Gær : Saccharomyces cerevisiae (bagergær)
Se også
- Liste over sekventerede eukaryote genomer
- Sekvensanalyse
- Sekvensprofileringsværktøj
- Sekvensmotiv
- UCSC Genome Browser
