Fagdisplay - Phage display
Fagdisplay er et laboratorium teknik til studiet af protein-protein , protein - peptid , og protein- DNA interaktioner, der bruger bakteriofager ( virus , der inficerer bakterier ) for at tilslutte proteiner med den genetiske information , som koder dem. Ved denne teknik er et gen der koder et protein af interesse indsættes i en fag- kappeprotein -gen, der forårsager fagen til "display" proteinet på sin yderside, mens indeholdende genet for proteinet på sin inderside, hvilket resulterer i en forbindelse mellem genotype og fænotype . Disse visningsfager kan derefter screenes mod andre proteiner, peptider eller DNA -sekvenser for at detektere interaktion mellem det viste protein og de andre molekyler. På denne måde kan store biblioteker af proteiner screenes og amplificeres i en proces kaldet in vitro -selektion, som er analog med naturlig selektion .
De mest almindelige bakteriofager, der bruges til fagvisning, er M13 og fd filamentøs fag , selvom T4 , T7 og λ fag også er blevet brugt.
Historie
Fagvisning blev først beskrevet af George P. Smith i 1985, da han demonstrerede visning af peptider på filamentøse fager (lange, tynde vira, der inficerer bakterier) ved at fusionere virusets kapsidprotein til et peptid ud af en samling af peptidsekvenser. Dette viste de forskellige peptider på de ydre overflader af samlingen af virale kloner, hvor screeningstrinnet i processen isolerede peptiderne med den højeste bindingsaffinitet. I 1988 beskrev Stephen Parmley og George Smith biopanning til valg af affinitet og demonstrerede, at rekursive selektionsrunder kunne berige for kloner, der er til stede på 1 ud af en milliard eller mindre. I 1990 beskrev Jamie Scott og George Smith oprettelsen af store tilfældige peptidbiblioteker, der blev vist på filamentøs fag. Phag -displayteknologi blev videreudviklet og forbedret af grupper på Laboratory of Molecular Biology med Greg Winter og John McCafferty , The Scripps Research Institute med Richard Lerner og Carlos Barbas og det tyske kræftforskningscenter med Frank Breitling og Stefan Dübel til visning af proteiner som f.eks. som antistoffer til terapeutisk proteinteknik . Smith og Winter blev tildelt en halv andel af Nobelprisen i kemi 2018 for deres bidrag til at udvikle fagvisning. Et patent af George Pieczenik, der hævder prioritet fra 1985, beskriver også frembringelsen af peptidbiblioteker.
Princip
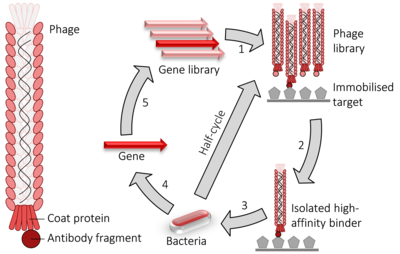
Ligesom to-hybridsystemet bruges fagdisplay til screening med høj gennemstrømning af proteininteraktioner. I tilfælde af M13 -filamentøs fagdisplay ligeres DNA'et, der koder for proteinet eller peptidet af interesse, ind i pIII- eller pVIII -genet, der koder for henholdsvis det mindre eller det store coatingsprotein . Flere kloningssteder bruges undertiden til at sikre, at fragmenterne indsættes i alle tre mulige læserammer, så cDNA -fragmentet translateres i den korrekte ramme. Faggenet og insert- DNA-hybrid indsættes derefter (en proces kendt som " transduktion ") i E. coli- bakterieceller, såsom TG1, SS320, ER2738 eller XL1-Blue E. coli . Hvis der bruges en " fagmid " -vektor (en forenklet displaykonstruktionvektor) vil fagpartikler ikke frigives fra E. coli -cellerne, før de er inficeret med hjælperfag , hvilket muliggør pakning af fag -DNA og samling af de modne virioner med relevante proteinfragment som en del af deres ydre pels på enten det mindre (pIII) eller det store (pVIII) pelsprotein. Ved at immobilisere et eller flere relevante DNA- eller proteinmål til overfladen af en mikrotiterpladebrønd , forbliver en fag, der viser et protein, der binder til et af disse mål på dens overflade, mens andre fjernes ved vask. De resterende kan elueres , bruges til at producere mere fag (ved bakteriel infektion med hjælperfag) og til at producere en fagblanding, der er beriget med relevant (dvs. bindende) fag. Den gentagne cyklus af disse trin omtales som 'panorering' med henvisning til berigelse af en prøve af guld ved at fjerne uønskede materialer. Fag elueret i det sidste trin kan bruges til at inficere en egnet bakteriel vært, hvorfra fagmiderne kan opsamles og den relevante DNA -sekvens udskæres og sekventeres for at identificere de relevante, interagerende proteiner eller proteinfragmenter.
Brugen af en hjælperfag kan elimineres ved hjælp af 'bakteriel emballage cellelinje' teknologi.
Eluering kan udføres kombinere lav pH eluering puffer med lydbehandling, som foruden at løsne peptid-target interaktion, også tjener til at frigøre målmolekylet fra immobiliseringen overflade. Denne ultralydsbaserede metode muliggør selektion i et trin af et peptid med høj affinitet.
Ansøgninger
Anvendelser af fagvisningsteknologi omfatter bestemmelse af interaktionspartnere for et protein (som ville blive brugt som den immobiliserede fag "agn" med et DNA -bibliotek bestående af alle kodende sekvenser af en celle, væv eller organisme), så funktionen eller mekanismen for proteinets funktion kan bestemmes. Fagvisning er også en meget udbredt metode til in vitro -proteinudvikling (også kaldet proteinteknik ). Som sådan er fagvisning et nyttigt værktøj til opdagelse af lægemidler . Det bruges til at finde nye ligander (enzymhæmmere, receptoragonister og antagonister) til at målrette proteiner. Teknikken bruges også til at bestemme tumorantigener (til brug ved diagnose og terapeutisk målretning) og i søgning efter protein-DNA-interaktioner ved hjælp af specialkonstruerede DNA-biblioteker med randomiserede segmenter. For nylig er fagvisning også blevet brugt i forbindelse med kræftbehandlinger - såsom adoptiv celleoverførselsmetode. I disse tilfælde bruges fagvisning til at oprette og vælge syntetiske antistoffer, der er målrettet mod tumoroverfladeproteiner. Disse bliver til syntetiske receptorer for T-celler indsamlet fra patienten, der bruges til at bekæmpe sygdommen.
Konkurrerende metoder til in vitro -proteinudvikling omfatter gærvisning , bakteriedisplay , ribosomvisning og mRNA -display .
Modning af antistof in vitro
Opfindelsen af antistoffagdisplay revolutionerede antistoflægemiddelopdagelse. Det første arbejde blev udført af laboratorier ved MRC Laboratory of Molecular Biology ( Greg Winter og John McCafferty ), Scripps Research Institute (Richard Lerner og Carlos F. Barbas) og det tyske kræftforskningscenter (Frank Breitling og Stefan Dübel). I 1991 rapporterede The Scripps -gruppen den første visning og selektion af humane antistoffer på fag. Denne indledende undersøgelse beskrev den hurtige isolering af humant antistof Fab, der bandt tetanustoksin, og metoden blev derefter udvidet til hurtigt at klone humane anti-HIV-1-antistoffer til vaccinedesign og terapi.
Fagvisning af antistofbiblioteker er blevet en kraftfuld metode til både at studere immunresponset samt en metode til hurtigt at udvælge og udvikle humane antistoffer til terapi. Antistof -fagdisplay blev senere brugt af Carlos F. Barbas ved The Scripps Research Institute til at oprette syntetiske humane antistofbiblioteker, et princip, der først blev patenteret i 1990 af Breitling og kolleger (Patent CA 2035384), hvorved menneskelige antistoffer kunne dannes in vitro fra syntetisk mangfoldighedselementer.
Antistofbiblioteker, der viser millioner af forskellige antistoffer på fag, bruges ofte i den farmaceutiske industri til at isolere meget specifikke terapeutiske antistofledninger til udvikling til antistoflægemidler primært som kræftbekæmpende eller antiinflammatoriske terapier. En af de mest succesrige var adalimumab , opdaget af Cambridge Antibody Technology som D2E7 og udviklet og markedsført af Abbott Laboratories . Adalimumab, et antistof mod TNF alpha , var verdens første fuldt humane antistof, der opnåede et årligt salg på over 1 mia.
Generel protokol
Nedenfor ses sekvensen af hændelser, der følges i fagvisningsscreening for at identificere polypeptider, der binder med høj affinitet til ønsket målprotein eller DNA -sekvens:
- Målproteiner eller DNA -sekvenser er immobiliseret til brøndene i en mikrotiterplade .
- Mange genetiske sekvenser udtrykkes i et bakteriofagbibliotek i form af fusioner med bakteriofagcoatproteinet, så de vises på overfladen af den virale partikel. Det viste protein svarer til den genetiske sekvens i fagen.
- Dette fagdisplaybibliotek tilføjes til fadet, og efter at fagetiden er blevet bundet, vaskes skålen.
- Fagdisplayende proteiner, der interagerer med målmolekylerne, forbliver knyttet til fadet, mens alle andre vaskes væk.
- Vedhæftet fag kan elueres og bruges til at skabe mere fag ved infektion af egnede bakterielle værter. Den nye fag udgør en beriget blanding, der indeholder betydeligt mindre irrelevant fag (dvs. ikke-bindende), end der var til stede i den oprindelige blanding.
- Trin 3 til 5 gentages eventuelt en eller flere gange, hvilket yderligere beriger fagbiblioteket i bindingsproteiner.
- Efter yderligere bakteriebaseret amplifikation sekventeres DNA inde i den interagerende fag til identifikation af de interagerende proteiner eller proteinfragmenter.
Udvælgelse af pelsproteinet
Filamentøse fager
pIII
pIII er proteinet, der bestemmer virions infektionsevne. pIII er sammensat af tre domæner (N1, N2 og CT) forbundet med glycinrige linkere. N2 -domænet binder sig til F pilus under virioninfektion, hvilket frigør N1 -domænet, som derefter interagerer med et TolA -protein på overfladen af bakterien. Insertioner i dette protein tilføjes sædvanligvis i position 249 (inden for en linkerregion mellem CT og N2), position 198 (inden for N2-domænet) og ved N-terminalen (indsat mellem den N-terminale sekretionssekvens og N-terminalen af pIII). Ved brug af BamHI-stedet i position 198 skal man dog være forsigtig med den uparede Cystein-rest (C201), der kan forårsage problemer under fagvisning, hvis man bruger en ikke-afkortet version af pIII.
En fordel ved at bruge pIII frem for pVIII er, at pIII giver mulighed for monovalent visning ved brug af et fagmid (plasmid afledt af Ff -fager ) kombineret med en hjælperfag. Desuden tillader pIII indsættelse af større proteinsekvenser (> 100 aminosyrer) og er mere tolerant over for det end pVIII. Imidlertid kan brug af pIII som fusionspartner føre til et fald i faginfektivitet, der kan føre til problemer såsom selektionsbias forårsaget af forskel i fagvæksthastighed eller endnu værre, fagets manglende evne til at inficere sin vært. Tab af faginfektivitet kan undgås ved at anvende et fagmidplasmid og en hjælperfag, så den resulterende fag indeholder både vildtype og fusionspIII.
cDNA er også blevet analyseret ved hjælp af pIII via et to komplementært leucin-lynlåssystem, Direct Interaction Rescue eller ved at tilføje en 8-10 aminosyrelinker mellem cDNA og pIII ved C-terminalen.
pVIII
pVIII er hovedkaffen protein af Ff fag. Peptider er normalt fusioneret til N-terminalen af pVIII. Normalt er peptider, der kan fusioneres til pVIII, 6-8 aminosyrer lange. Størrelsesbegrænsningen synes at have mindre at gøre med strukturel hindring forårsaget af den tilføjede sektion og mere at gøre med størrelsesekskluderingen forårsaget af pIV under eksport af pelsprotein. Da der er omkring 2700 kopier af proteinet på en typisk fag, er det mere sandsynligt, at proteinet af interesse vil blive udtrykt polyvalent, selvom der bruges et fagemid. Dette gør brugen af dette protein ugunstig til opdagelse af bindingspartnere med høj affinitet.
For at overvinde størrelsesproblemet med pVIII er kunstige coatingsproteiner designet. Et eksempel er Weiss og Sidhu's inverterede kunstige kappeprotein (ACP), som tillader visning af store proteiner ved C-terminalen. ACP'erne kunne dog vise et protein på 20kDa, dog kun ved lave niveauer (for det meste kun monovalent).
pVI
pVI er blevet udbredt til visning af cDNA -biblioteker. Visningen af cDNA-biblioteker via fagdisplay er et attraktivt alternativ til gær-2-hybridmetoden til opdagelse af interagerende proteiner og peptider på grund af dets høje kapacitet. pVI er fortrinsvis blevet brugt til pVIII og pIII til ekspression af cDNA-biblioteker, fordi man kan tilføje proteinet af interesse til C-terminalen af pVI uden i høj grad at påvirke pVI's rolle i fagsamling. Dette betyder, at stopkodonet i cDNA ikke længere er et problem. Imidlertid er fagvisning af cDNA altid begrænset af de fleste prokaryotes manglende evne til at producere posttranslationelle modifikationer til stede i eukaryote celler eller ved fejlfoldning af multi-domæne proteiner.
Selvom pVI har været nyttig til analyse af cDNA -biblioteker, forbliver pIII og pVIII de mest anvendte coatingsproteiner til fagvisning.
pVII og pIX
I et forsøg i 1995 blev visning af Glutathione S-transferase forsøgt på både pVII og pIX og mislykkedes. Imidlertid blev fagvisning af dette protein afsluttet med succes efter tilføjelsen af en periplasmatisk signalsekvens (pelB eller ompA) på N-terminalen. I en nylig undersøgelse er det blevet vist, at AviTag, FLAG og His kunne vises på pVII uden behov for en signalsekvens. Derefter blev ekspressionen af enkeltkædede Fv'er (scFv) og enkeltkædede T -celleceptorer (scTCR) udtrykt både med og uden signalsekvensen.
PelB (en aminosyresignalsekvens, der målretter proteinet mod periplasmaet, hvor en signalpeptidase derefter spaltes af PelB) forbedrede fagvisningsniveauet sammenlignet med pVII- og pIX -fusioner uden signalsekvensen. Dette førte imidlertid til inkorporering af flere hjælper -fag -genomer frem for fag -mid -genomer. I alle tilfælde var fagvisningsniveauer lavere end ved brug af pIII -fusion. Imidlertid kan lavere display være mere gunstigt for valg af bindemidler på grund af lavere display, der er tættere på ægte monovalent display. I fem ud af seks lejligheder var pVII- og pIX -fusioner uden pelB mere effektive end pIII -fusioner i affinitetsvalgassays. Papiret fortsætter endda med at konstatere, at pVII- og pIX -displayplatforme kan overgå pIII i det lange løb.
Brugen af pVII og pIX i stedet for pIII kan også være en fordel, fordi virion-redning kan foretages uden at bryde virion-antigenbindingen, hvis den anvendte pIII er vildtype. I stedet kunne man kløve i et snit mellem perlen og antigenet for at eluere. Da pIII er intakt, er det ligegyldigt, om antigenet forbliver bundet til fagen.
T7 fager
Spørgsmålet om brug af Ff -fager til fagvisning er, at de kræver, at proteinet af interesse translokeres hen over den bakterielle indre membran, før de samles i fagen. Nogle proteiner kan ikke gennemgå denne proces og kan derfor ikke vises på overfladen af Ff -fager. I disse tilfælde bruges T7 -fagdisplay i stedet. I T7-fagdisplay er proteinet, der skal vises, knyttet til C-terminalen af gen 10-capsidproteinet i T7.
Ulempen ved at bruge T7 er, at størrelsen af det protein, der kan udtrykkes på overfladen, er begrænset til kortere peptider, fordi store ændringer i T7 -genomet ikke kan rummes, som det er i M13, hvor fagen bare gør pelsen længere for at passe til større genom indeni den. Det kan imidlertid være nyttigt til fremstilling af et stort proteinbibliotek til scFV -selektion, hvor scFV udtrykkes på en M13 -fag, og antigenerne udtrykkes på overfladen af T7 -faget.
Bioinformatik ressourcer og værktøjer
Databaser og beregningsværktøjer til mimotoper har været en vigtig del af fagdisplaystudiet. Databaser, programmer og webservere er blevet udbredt til at udelukke målrelaterede peptider, karakterisere små molekyler-protein-interaktioner og kortlægge protein-protein-interaktioner. Brugere kan bruge tredimensionel struktur af et protein og peptiderne valgt fra fagdisplayeksperiment til at kortlægge konformationelle epitoper. Nogle af de hurtige og effektive beregningsmetoder er tilgængelige online.
Se også
Konkurrerende teknikker:
Referencer
Yderligere læsning
- Ledsgaard L, Kilstrup M, Karatt-Vellatt A, McCafferty J, Laustsen AH (2018). "Grundlæggende om antistoffagvisningsteknologi" (PDF) . Toksiner . 10 (6): 236. doi : 10.3390/toksiner10060236 . PMC 6024766 . PMID 29890762 .
- Udvælgelse mod design i kemiteknik
- ETH-2 humant antistof fagbibliotek
- Sidhu SS, Lowman HB, Cunningham BC, Wells JA (2000). "Fagdisplay til udvælgelse af nye bindende peptider". Meth. Enzymol . Metoder i enzymologi. 328 : 333–63. doi : 10.1016/S0076-6879 (00) 28406-1 . ISBN 9780121822293. PMID 11075354 .
eksterne links
|
Biblioteksressourcer om fagvisning |
