Zink finger - Zinc finger

En zinkfinger er en lille protein strukturelt motiv , som er karakteriseret ved koordinering af et eller flere zink -ioner (Zn 2+ ) for at stabilisere folden. Det blev oprindeligt opfundet til at beskrive det fingerlignende udseende af en hypotetiseret struktur fra den afrikanske klofrø ( Xenopus laevis ) transkriptionsfaktor IIIA . Det har imidlertid vist sig at omfatte en lang række forskellige proteinstrukturer i eukaryote celler. Xenopus laevis TFIIIA blev oprindeligt påvist at indeholde zink og kræver metallet til funktion i 1983, det første sådanne rapporterede zinkbehov for et genregulerende protein efterfulgt kort derefter af Krüppel -faktoren i Drosophila . Det fremstår ofte som et metalbindende domæne i multi-domæne proteiner.
Proteiner, der indeholder zinkfingre ( zinkfingerproteiner ), er klassificeret i flere forskellige strukturelle familier. I modsætning til mange andre klart definerede supersekundære strukturer såsom græske nøgler eller β-hårnåle , er der en række typer zinkfingre med hver en unik tredimensionel arkitektur. Et bestemt zinkfingerproteins klasse bestemmes af denne tredimensionelle struktur, men det kan også genkendes baseret på proteinets primære struktur eller identiteten af liganderne, der koordinerer zinkionen. På trods af den store variation af disse proteiner fungerer langt de fleste typisk imidlertid som interaktionsmoduler, der binder DNA , RNA , proteiner eller andre små nyttige molekyler, og variationer i struktur tjener primært til at ændre bindingsspecificiteten af et bestemt protein .
Siden deres oprindelige opdagelse og belysning af deres struktur har disse interaktionsmoduler vist sig allestedsnærværende i den biologiske verden og kan findes i 3% af generne i det menneskelige genom. Derudover er zinkfingre blevet yderst nyttige i forskellige terapeutiske og forskningskapaciteter. Engineering af zinkfingre til at have en affinitet for en bestemt sekvens er et område med aktiv forskning, og zinkfingernukleaser og zinkfinger -transkriptionsfaktorer er to af de vigtigste anvendelser heraf, der skal realiseres til dato.
Historie
Zinkfingre blev først identificeret i en undersøgelse af transkription i den afrikanske klofrø , Xenopus laevis i Aaron Klugs laboratorium . En undersøgelse af transkriptionen af en bestemt RNA-sekvens afslørede, at bindingsstyrken af en lille transkriptionsfaktor (transkriptionsfaktor IIIA; TFIIIA) skyldtes tilstedeværelsen af zink-koordinerende fingerlignende strukturer. Aminosyresekventering af TFIIIA afslørede ni tandemsekvenser af 30 aminosyrer, herunder to invariante par cystein- og histidinrester. Udvidet røntgenabsorptionsfinstruktur bekræftede zinkligandernes identitet: to cystein og to histidiner. Den DNA-bindende sløjfe dannet ved koordinering af disse ligander med zink blev antaget at ligne fingre, deraf navnet. Dette blev fulgt kort derefter med opdagelsen af Krüppel -faktoren i Drosophila af Schuh -teamet i 1986. Nyere arbejde med karakterisering af proteiner i forskellige organismer har afsløret betydningen af zinkioner ved polypeptidstabilisering.
Krystalstrukturerne af zinkfinger-DNA-komplekser løst i 1991 og 1993 afslørede det kanoniske mønster af interaktioner mellem zinkfingre og DNA. Bindningen af zinkfingeren viser sig at være forskellig fra mange andre DNA-bindende proteiner, der binder DNA gennem den dobbelte helix 2-symmetri, i stedet er zinkfingre forbundet lineært i tandem for at binde nukleinsyresekvenser af varierende længder. Zinkfingre binder ofte til en DNA -sekvens kendt som GC -boksen . Zinkfingermotivets modulære karakter gør det muligt for et stort antal kombinationer af DNA- og RNA -sekvenser at blive bundet med høj grad af affinitet og specificitet og er derfor ideelt egnet til konstruktion af protein, der kan målrettes til og binde specifikke DNA -sekvenser. I 1994 blev det vist, at et kunstigt konstrueret trefingers protein kan blokere ekspressionen af et onkogen i en musecellelinje. Zinkfingre smeltet til forskellige andre effektordomæner, nogle med terapeutisk betydning, er siden blevet konstrueret.
Domæne
Zinkfinger (Znf) domæner er relativt små proteinmotiver, der indeholder flere fingerlignende fremspring, der skaber tandemkontakter med deres målmolekyle. Nogle af disse domæner binder zink, men mange binder det ikke i stedet for andre metaller såsom jern eller slet ikke metal. For eksempel danner nogle familiemedlemmer saltbroer for at stabilisere de fingerlignende folder . De blev først identificeret som en DNA-bindende motiv i transskriptionsfaktor TFIIIA fra Xenopus laevis (Xenopus laevis), men de er nu anerkendt at binde DNA, RNA, protein og / eller lipid substrater . Deres bindingsegenskaber afhænger af aminosyresekvensen af fingerdomænerne og af linkeren mellem fingre samt af strukturer af højere orden og antallet af fingre. Znf -domæner findes ofte i klynger, hvor fingre kan have forskellige bindingsspecificiteter. Znf -motiver forekommer i flere ikke -relaterede proteinsuperfamilier , der varierer i både sekvens og struktur. De udviser betydelig alsidighed i bindingsmetoder, selv mellem medlemmer af samme klasse (f.eks. Nogle binder DNA, andre proteiner), hvilket tyder på, at Znf -motiver er stabile stilladser, der har udviklet specialiserede funktioner. For eksempel Znf-holdige proteiner fungerer i gen transkription, translation, mRNA handel, cytoskeleton organisation, epitelial udvikling, celleadhæsion , proteinfoldning, chromatin remodeling, og zink sensing, for at nævne nogle få. Zinkbindende motiver er stabile strukturer, og de undergår sjældent konformationsændringer ved binding af deres mål.
Klasser
Oprindeligt blev udtrykket zinkfinger udelukkende brugt til at beskrive DNA-bindende motiv fundet i Xenopus laevis ; den bruges imidlertid nu til at referere til et hvilket som helst antal strukturer, der er forbundet med deres koordinering af en zinkion. Generelt koordinerer zinkfingre zinkioner med en kombination af cystein- og histidinrester . Oprindeligt blev antallet og rækkefølgen af disse rester brugt til at klassificere forskellige typer zinkfingre (f.eks. Cys 2 His 2 , Cys 4 og Cys 6 ). For nylig er en mere systematisk metode blevet brugt til at klassificere zinkfingerproteiner i stedet. Denne metode klassificerer zinkfingerproteiner i "foldgrupper" baseret på den overordnede form af proteinets rygrad i det foldede domæne. De mest almindelige "foldgrupper" af zinkfingre er Cys 2 His 2 -lignende (den "klassiske zinkfinger"), diskantnøgle og zinkbånd.
Følgende tabel viser de forskellige strukturer og deres nøglefunktioner:
Cys 2 His 2
| Zinkfinger, type C2H2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identifikatorer | |||||||||
| Symbol | zf-C2H2 | ||||||||
| Pfam | PF00096 | ||||||||
| Pfam klan | CL0361 | ||||||||
| InterPro | IPR007087 | ||||||||
| PROSIT | PS00028 | ||||||||
| |||||||||
Cys 2 His 2 -lignende fold -gruppen (C2H2) er langt den bedst karakteriserede klasse zinkfingre og er almindelig i pattedyrstranskriptionsfaktorer. Sådanne domæner vedtage en enkel ββα fold og har aminosyresekvensen sekvensmotiv :
- X 2 -Cys -X 2,4 -Cys -X 12 -His -X 3,4,5 -His
Denne klasse af zinkfingre kan have en række funktioner, såsom at binde RNA og mediere protein-protein-interaktioner, men er bedst kendt for sin rolle i sekvens-specifikke DNA-bindende proteiner, såsom Zif268 (EGR1). I sådanne proteiner forekommer individuelle zinkfingerdomæner typisk som tandem-gentagelser med to, tre eller flere fingre omfattende proteinets DNA-bindende domæne. Disse tandem-arrays kan binde i den store rille af DNA og er typisk i afstand med 3-bp intervaller. A-helixen for hvert domæne (ofte kaldet "genkendelsesspiralen") kan oprette sekvensspecifikke kontakter til DNA-baser; rester fra en enkelt genkendelsesspiral kan komme i kontakt med fire eller flere baser for at give et overlappende mønster af kontakter med tilstødende zinkfingre.
Gag-kno
| Zink kno | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identifikatorer | |||||||||
| Symbol | zf-CCHC | ||||||||
| Pfam | PF00098 | ||||||||
| InterPro | IPR001878 | ||||||||
| SMART | SM00343 | ||||||||
| PROSIT | PS50158 | ||||||||
| |||||||||
Denne foldgruppe er defineret af to korte β-tråde forbundet med en drejning (zink-kno) efterfulgt af en kort spiral eller sløjfe og ligner det klassiske Cys 2 His 2- motiv med en stor del af spiralen og β-hårnåle afkortet.
Det retrovirale nucleocapsid (NC) protein fra HIV og andre beslægtede retrovira er eksempler på proteiner, der besidder disse motiver. Gag-knuckle zinkfingeren i HIV NC-proteinet er målet for en klasse af lægemidler kendt som zinkfingerhæmmere .
G-nøgle
Treble-clef-motivet består af en β-hårnål ved N-terminalen og en α-helix ved C-terminalen, der hver bidrager med to ligander til zinkbinding, selvom en loop og en anden β-hårnål af varierende længde og konformation kan være til stede mellem den N-terminale β-hårnål og den C-terminale α-helix. Disse fingre er til stede i en forskelligartet gruppe af proteiner, der ofte ikke deler sekvens eller funktionel lighed med hinanden. De bedst karakteriserede proteiner indeholdende diskant-nøgle zinkfingre er de nukleare hormonreceptorer .
Zinkbånd
| TFIIB zinkbinding | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identifikatorer | |||||||||
| Symbol | TF_Zn_Ribbon | ||||||||
| Pfam | PF08271 | ||||||||
| Pfam klan | Zn_Beta_Ribbon | ||||||||
| InterPro | IPR013137 | ||||||||
| PROSIT | PS51134 | ||||||||
| |||||||||
Zinkbåndfolden er kendetegnet ved to beta-hårnåle, der danner to strukturelt ensartede zinkbindende undersider.
Zn 2 /Cys 6
| Svampe Zn (2) -Cys (6) binuklear klynge domæne | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identifikatorer | |||||||||
| Symbol | Zn_clus | ||||||||
| Pfam | PF00172 | ||||||||
| InterPro | IPR001138 | ||||||||
| SMART | GAL4 | ||||||||
| PROSIT | PS00463 | ||||||||
| CDD | cd00067 | ||||||||
| |||||||||
De kanoniske medlemmer af denne klasse indeholder en binuklear zinkhob, hvor to zinkioner er bundet af seks cysteinrester . Disse zinkfingre kan findes i flere transkriptionsfaktorer, herunder gær Gal4 -proteinet.
| zf-C2HC | |||||||||
|---|---|---|---|---|---|---|---|---|---|
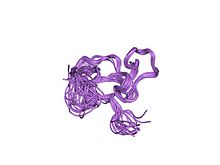 opløsningsstruktur for et cchhc-domæne af neuralt zinkfingerfaktor-1
| |||||||||
| Identifikatorer | |||||||||
| Symbol | zf-C2HC | ||||||||
| Pfam | PF01530 | ||||||||
| InterPro | IPR002515 | ||||||||
| |||||||||
| zf-C2HC5 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identifikatorer | |||||||||
| Symbol | zf-C2HC5 | ||||||||
| Pfam | PF06221 | ||||||||
| InterPro | IPR009349 | ||||||||
| |||||||||
Diverse
De zinkfinger antiviralt protein (ZAP) binder til CpG stedet. Det bruges i pattedyr til antiviralt forsvar.
Ansøgninger
Forskellige proteintekniske teknikker kan bruges til at ændre zinkfingers DNA-bindingsspecificitet, og tandem-gentagelser af sådanne manipulerede zinkfingre kan bruges til at målrette ønskede genomiske DNA-sekvenser. Fusionering af et andet proteindomæne, såsom en transkriptionel aktivator eller repressor til en række manipulerede zinkfingre, der binder nær promotoren for et givet gen, kan bruges til at ændre transskriptionen af det gen. Fusioner mellem konstruerede zinkfingerarrays og proteindomæner, der spalter eller på anden måde modificerer DNA, kan også bruges til at målrette disse aktiviteter mod ønskede genomiske loci. De mest almindelige anvendelser for konstruerede zinkfingerarrays omfatter zinkfinger -transkriptionsfaktorer og zinkfingernukleaser , men andre applikationer er også blevet beskrevet. Typiske konstruerede zinkfingerarrays har mellem 3 og 6 individuelle zinkfingermotiver og binder målsteder, der spænder fra 9 basepar til 18 basepar i længden. Arrays med 6 zinkfingermotiver er særligt attraktive, fordi de binder et målsted, der er langt nok til at have en god chance for at være unik i et pattedyrs genom.
Zinkfingernukleaser
Konstruerede zinkfingerarrays er ofte fusioneret til et DNA -spaltningsdomæne (normalt FokI 's spaltningsdomæne ) for at generere zinkfingernukleaser . Sådanne zinkfinger-FokI-fusioner er blevet nyttige reagenser til manipulation af genomer af mange højere organismer, herunder Drosophila melanogaster , Caenorhabditis elegans , tobak , majs , zebrafisk , forskellige typer pattedyrsceller og rotter . Målretning af en dobbeltstrengsbrud til et ønsket genomisk locus kan bruges til at indføre rammeskiftmutationer i et kodende sekvens af et gen på grund af den fejl-tilbøjelige natur af den ikke-homologe DNA-reparationsvej. Hvis der også anvendes en homolog DNA "donorsekvens", kan det genomiske locus konverteres til en defineret sekvens via den homologirettede reparationsvej. Et igangværende klinisk forsøg evaluerer zinkfingernukleaser, der forstyrrer CCR5-genet i CD4 + humane T-celler som en potentiel behandling for HIV/AIDS .
Metoder til konstruktion af zinkfingerarrays
Størstedelen af konstruerede zinkfingerarrays er baseret på zinkfingerdomænet for den murine transkriptionsfaktor Zif268, selvom nogle grupper har brugt zinkfingerarrays baseret på den humane transkriptionsfaktor SP1. Zif268 har tre individuelle zinkfingermotiver, der samlet binder en 9 bp sekvens med høj affinitet. Strukturen af dette protein, der er bundet til DNA, blev løst i 1991 og stimulerede megen forskning i konstruerede zinkfingerarrays. I 1994 og 1995 brugte en række grupper fagvisning til at ændre specificiteten af en enkelt zinkfinger af Zif268. Der er i øjeblikket to hovedmetoder, der bruges til at generere manipulerede zinkfingerarrays, modulopbygning og et bakterieselektionssystem, og der er en vis debat om, hvilken metode der er bedst egnet til de fleste applikationer.
Den mest enkle metode til at generere nye zinkfingerarrays er at kombinere mindre zinkfinger "moduler" med kendt specificitet. Strukturen af zinkfingerproteinet Zif268 bundet til DNA beskrevet af Pavletich og Pabo i deres publikation fra 1991 har været nøglen til meget af dette arbejde og beskriver konceptet med at få fingre til hver af de 64 mulige basepar -trillinger og derefter blande og matche disse fingre til at designe proteiner med en hvilken som helst ønsket sekvensspecificitet. Den mest almindelige modulære samlingsproces indebærer at kombinere separate zinkfingre, der hver kan genkende en 3-basepar DNA-sekvens for at generere 3-finger, 4-, 5- eller 6-finger-arrays, der genkender målsteder fra 9 basepar til 18 basepar i længde. En anden metode bruger 2-finger moduler til at generere zink finger arrays med op til seks individuelle zink fingre. Barbas Laboratory fra The Scripps Research Institute brugte fagvisning til at udvikle og karakterisere zinkfingerdomæner, der genkender de fleste DNA -tripletsekvenser, mens en anden gruppe isolerede og karakteriserede individuelle fingre fra det menneskelige genom. En potentiel ulempe ved modulopbygning generelt er, at specificiteter af individuelle zinkfingre kan overlappe hinanden og kan afhænge af konteksten for de omgivende zinkfingre og DNA. En nylig undersøgelse viste, at en stor andel af 3-finger zinkfingerarrays genereret af modulær samling ikke binder deres tilsigtede mål med tilstrækkelig affinitet i et bakterielt to-hybrid assay og ikke fungerer som zinkfingernukleaser , men succesraten var noget højere, når websteder med formen GNNGNNGNN blev målrettet.
En efterfølgende undersøgelse anvendte modulær samling til at generere zinkfinger-nukleaser med både 3-finger-arrays og 4-finger-arrays og observerede en meget højere succesrate med 4-finger-arrays. En variant af modulopbygning, der tager konteksten med nabofingre i betragtning, er også blevet rapporteret, og denne metode har en tendens til at give proteiner med forbedret ydeevne i forhold til standard modulopbygning.
Talrige selektionsmetoder er blevet brugt til at generere zinkfingerarrays, der er i stand til at målrette mod ønskede sekvenser. Indledende selektionsbestræbelser anvendte fagdisplay til at vælge proteiner, der bandt et givet DNA -mål fra en stor pulje af delvist randomiserede zinkfingerarrays. Denne teknik er vanskelig at bruge på mere end en enkelt zinkfinger ad gangen, så der blev udviklet en proces i flere trin, der genererede et fuldstændigt optimeret 3-finger-array ved at tilføje og optimere en enkelt zinkfinger ad gangen. Nyere indsats har anvendt gær-en-hybridsystemer, bakterielle en-hybrid og to-hybrid systemer og pattedyrceller. En lovende ny metode til at vælge nye 3-finger zink finger-arrays anvender et bakterielt to-hybrid system og er blevet kaldt "OPEN" af dets skabere. Dette system kombinerer forudvalgte puljer af individuelle zinkfingre, der hver især blev valgt til at binde en given triplet og anvender derefter en anden valgrunde til at opnå 3-finger-arrays, der er i stand til at binde en ønsket 9-bp-sekvens. Dette system blev udviklet af Zinkfinger -konsortiet som et alternativ til kommercielle kilder til konstruerede zinkfingerarrays. Det er noget svært at direkte sammenligne bindingsegenskaberne for proteiner, der genereres med denne metode, til proteiner, der genereres ved modulær samling, da specificitetsprofiler for proteiner, der genereres ved OPEN -metoden, aldrig er blevet rapporteret.
Eksempler
Denne post repræsenterer CysCysHisCys (C2HC) zinkfingerdomæne fundet i eukaryoter . Proteiner, der indeholder disse domæner, omfatter:
- MYST -familie histonacetyltransferaser
- Myelin -transkriptionsfaktor Myt1
- Undertrykker af tumorigenicitetsprotein 18 (ST18)
Se også
Referencer
eksterne links
- C2H2 familie Arkiveret 2016-02-15 på Wayback Machine på PlantTFDB: Plant Transcription Factor Database
- McDowall J. "Månedens protein: zinkfingre" . European Molecular Biology Laboratory - European Bioinformatics Institute (EMBL -EBI) . Hentet 2008-01-13 .
- Varesalg DS. "Månedens molekyle: zinkfingre" . Research Collaboratory for Structural Bioinformatics (RCSB) Protein Data Bank (PDB). Arkiveret fra originalen 2008-05-31 . Hentet 2008-01-13 .
- Den dobbelte spiral mellem zinkfingeren
- Zinkfingerværktøjs design- og informationswebsted
- Human KZNF Gene Catalog Archived 2012-03-16 at Wayback Machine
- Zinkfinger C2H2-type domæne i PROSITE
- Indgang til zinkfingerklasse C2H2 i SMART -databasen
- Zinkfinger -konsortiet
- ZiFiT- design af værktøj til zinkfinger
- Zink Finger Consortium Materials fra Addgene
- Forudsiger DNA-bindende specificiteter for C2H2 zinkfingerproteiner